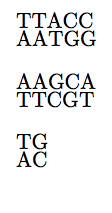字體:小 中 大
字體:小 中 大 |
|
|
|
| 2013/07/16 07:29:58瀏覽324|回應0|推薦1 | |
%這次練習字串運算,設計宏來進行字串的運算與轉換。DNA主要是由四種氮基結構的配對組成,A、T、C、G四種,而雙股DNA則是A和T配對,C和G配對。而現在就設計宏把單股DNA序列轉成它的配對序列,並且配對成雙股DNA序列。 def antiString (expr t) = begingroup save s, antiString; string s[], antiString; antiString="" for i=0 upto length t: hide( s[i]=substring(i,i+1) of t; if s[i]="A": s[i]:="T"; elseif s[i]="T": s[i]="A"; elseif s[i]="C": s[i]="G"; elseif s[i]="G": s[i]="C"; fi ) &s[i] endfor ; antiString endgroup enddef; %字串的相加是用&。&前後都要有字串,如果沒有字串,那用空字串""就可以。hide的內容在前面的文章介紹過,它是一個不會為真的判斷式。但判斷式的內容會運算過一遍,而運算結果就直接可以使用。所以antiString="" for i=0 utpo length t: hide( .......) &s[i] endfor ; 的運算結果就是 antiString = "" &s0&s1&s2&s3...; s[i]的內容在hide()內已算過一遍了,所以不會影響字串的相加運算。最後運算結果就是antiString的值。 %接下來畫出雙股DNA: def draw_dsDNA(expr t)= begingroup save ssDNA, asDNA; string ssDNA[], asDNA[]; if length t > 5:%檢查字串長度 for i=0 upto floor (length t -1)/5:%分別截取長度為5的字串 ssDNA[i]=substring(5*i, 5*i+5) of t; asDNA[i]=antiString(ssDNA[i]); label.rt(ssDNA[i],(0,-i*cm)); labe.rt(asDNA[i],(0,(-i-0.3)*cm)); endfor else: ssDNA0=t; asDNA0=antiString(t); label.rt(ssDNA0,(0,0)); label.rt(asDNA0,(0,-0.3cm)); fi endgroup enddef; %draw_dsDNA也很簡單,主要就是把二個字串用label指令畫出來。因為考慮到DNA序列很長,所以在5個DNA氮基長度就把它切斷。這個字串要表示多長則可以視需要改變其中判斷式的內容。 %接下來就是實際上畫出來: beginfig(1); string a; a="TAACCAAGCATG" draw_dsDNA(a); endfig;
%其他的運算,如檢查EcoR1的位置,可以用 pair position[]; def position_Ecor1_check(expr t) = begingroup save check; check:=0; for i=0 upto length t: if substring (i, i+6) of t = "GAATTC": position[check]=(i,i+6); check:=check+1; fi endfor if check=0: show "No EcoR1 site!"; fi endgroup; enddef; %position[]的內容就是EcoR1在DNA字串中的位置,然後再視需要再做出運算。DNA的序列很長,可以用readfom "fileName" 取得檔案內一行字串內容。 |
|
| ( 興趣嗜好|電腦3C ) |